Epigenética: candados y llaves durante la lectura del ADN
Vol. 21, núm. 6 noviembre-diciembre 2020
Epigenética: candados y llaves durante la lectura del ADN
María Teresa Arceo-Martínez, Viviana Valadez-Graham y Zoraya Palomera-Sanchez CitaResumen
Hasta mediados del siglo pasado, se pensaba que el desarrollo y las características fenotípicas de los organismos dependían únicamente de la información comprendida en los genes. Asimismo, la propensión a enfermedades se atribuía únicamente a mutaciones en la secuencia de adn de los genes. No obstante, en las últimas décadas se ha puesto especial atención a un nuevo tipo de regulación genómica en la célula: la epigenética. Esta rama de la biología estudia cómo los factores externos de la célula regulan la expresión de los genes sin modificar su secuencia de nucleótidos. Dicha regulación se basa en cambios químicos que pueden actuar como llaves o candados para permitir o bloquear la expresión de un gen. De manera interesante, el ambiente constituye el principal modulador epigenético, ya que, dependiendo de las condiciones a las que esté expuesto el organismo, determinará cuáles genes se abren y cuáles se bloquean. Así, lo que comemos, lo que percibimos y el lugar en donde vivimos, regularán la forma en que se expresan nuestros genes a lo largo de nuestra vida.
Palabras clave: genética, epigenética, expresión de genes, ambiente, desarrollo, enfermedades.
Epigenetics: padlocks and keys during DNA reading
Abstract
Until the middle of the last century, we knew that the development and the phenotypic characteristics of organisms depended on the dna sequence of genes. Also, propensity/susceptibility to illness were only attributed to changes in the dna sequence. Nevertheless, in the last decades special attention has been paid to a new type of regulation in the cell: epigenetics. This branch of biology studies how the external factors regulate gene expression without affecting the nucleotide sequence. This regulation is based on the chemical groups that can act as keys or padlocks to open or block gene expression. Interestingly, the environment is the main epigenetic regulator, because external conditions influence which genes are locked or opened for expression. Therefore, the food we eat, things we perceive and where we live, regulate the way genes are expressed throughout our lives.
Keywords: genetic, epigenetic, gene expression, environment, development, illness.
Introducción
¿Te has preguntado alguna vez por qué los gemelos idénticos, a pesar de que tienen la misma información genética pueden ser diferentes entre ellos? Algunos difieren en la complexión, otros en el tono de piel o inclusive en la susceptibilidad a enfermedades.
Cada célula de nuestro cuerpo funciona a través de una serie de instrucciones que se guarda en forma de genes, muchos de los cuales dan origen a proteínas responsables de la estructura, color, función, etcétera, de los tejidos de los seres vivos. En los gemelos, este proceso es idéntico, por lo que conservan el mismo patrón de apagado y encendido de genes hasta el momento de nacer, cuando se enfrentan al ambiente. Conforme crecen y envejecen, se exponen a diversas condiciones ambientales, por lo que cada uno desarrolla diferentes patrones en la expresión de genes (Fraga et al., 2005).
En décadas pasadas, la respuesta a la pregunta de las diferencias físicas entre gemelos, si no también la propensión a enfermedades se había basado en la genética, es decir, que dichos cambios se explicaban debido a la acumulación de alteraciones (mutaciones) en la secuencia de adn, de cada uno de los hermanos a lo largo de la vida. No obstante, en los últimos años, se ha visto que existe otro fenómeno que altera la expresión de los genes sin modificar la secuencia de adn y que está modulado por las condiciones ambientales: se trata de un conjunto de eventos explicados por la epigenética. En un estudio de gemelos de la nasa, un gemelo vivió en el espacio por un año, y presentó cambios epigenéticos (en los patrones de metilación del adn), que modularon diferentes eventos celulares como el acortamiento en la longitud de los telómeros, cambios en la expresión de genes (del sistema inmune, metabólico, cardiovascular) y daño al adn; en comparación con el gemelo que permaneció en la tierra (Garrett-Bakelman et al., 2019; ver figura 1).

Figura 1. La epigenética explica las diferencias en el fenotipo de los gemelos idénticos. Estudios de la nasa con los gemelos idénticos Scott and Mark Kelly muestran cambios en el fenotipo después de que uno de ellos permaneció en el espacio por un año (Scott, derecha), mientras que el otro permaneció en la tierra como sujeto control (Mark, izquierda). Imagen tomada de https://www.nasa.gov/feature/nasa-twins-study-confirms-preliminary-findings.
La genética y la epigenética: la biblioteca celular
Pero… ¿qué es la epigenética? Esta palabra significa “sobre la genética” y fue utilizada por primera vez en 1942 por Conrad Waddintong, quien la definió, para explicar aspectos del desarrollo, como los cambios que se dan en el fenotipo, sin presentar cambios en el genotipo (Waddington, 1942). Actualmente se entiende como los cambios químicos que alteran la expresión genética, sin modificar la secuencia del adn (Allis et al., 2015).
Para entender mejor la diferencia entre genética y epigenética, podemos hacer una analogía entre la célula y una fábrica. Dentro de la célula, el núcleo contiene la cromatina, la cual es la plataforma constituida por la unión del adn, histonas y otras proteínas. Las histonas son proteínas sobre las que se enrolla el adn para organizarlo y compactarlo. El adn constituye los genes, que tienen una secuencia específica de nucleótidos,1 la cual dicta las instrucciones para el funcionamiento celular (Allis et al., 2015). Volviendo a la analogía, dentro de la fábrica existe una biblioteca (núcleo celular), que contiene todas las instrucciones para su funcionamiento, y éstas se encuentran ordenadas en salas de lectura con plataformas que las sostienen (cromatina); dichas plataformas se organizan en estantes (histonas), los cuales tienen libros (adn-genes). Cada libro o gen contiene una secuencia de palabras que son leídas por operadores especializados (proteínas). Si la secuencia cambia (por ejemplo, por una mutación), el operador no entenderá las instrucciones o los leerá de manera errónea y el proceso no se llevará a cabo o se hará de forma incorrecta (figura 2).
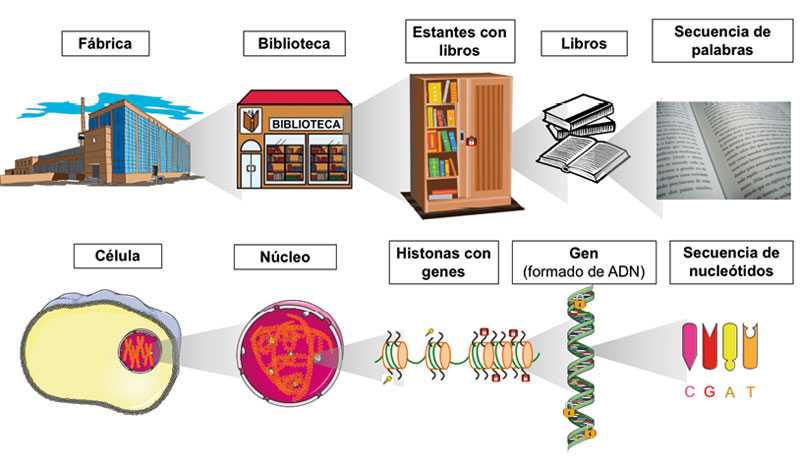
Figura 2. El funcionamiento de la célula se asemeja al de una fábrica. Dentro de ella hay una biblioteca llena de libros que contiene todas las instrucciones necesarias para su funcionamiento. En la célula, el núcleo es quien almacena el adn donde se encuentra toda la información para el funcionamiento celular. Los símbolos de llaves y candados en las histonas y candados en el adn representan las modificaciones epigenéticas que regulan la actividad o el bloqueo de la expresión de secuencias de adn, como los genes.
En la organización de la biblioteca, hay salas de lectura que tienen acceso restringido y se localizan en los lugares más recónditos del edificio. También hay áreas especiales que se usan ocasionalmente o bajo circunstancias específicas. Finalmente, los libros que se requieren en el día a día están acomodados en las salas principales, las cuales son fácilmente accesibles para la mayoría de los consultores.
De igual forma sucede en el núcleo celular: dentro de la cromatina hay secuencias de adn que pueden ser letales para la estabilidad del genoma (como retrovirus insertados), por lo que se ubican en regiones muy compactas, de difícil acceso y ubicadas generalmente en la periferia nuclear (heterocromatina constitutiva); otras secuencias de adn o genes que se expresan ocasionalmente o bajo circunstancias específicas están en regiones medianamente compactas en el cuerpo del núcleo (heterocromatina facultativa). Finalmente, hay un grupo de genes ubicados en zonas de fácil acceso, con un empaquetamiento laxo que permite su expresión constante (eucromatina). Aquí se encuentran los genes que se expresan día con día para el funcionamiento básico de la célula (Allis et al., 2015; Allis y Jenuwein, 2016).
Adicionalmente, muchos libros de consulta temporal o prohibidos se encuentran acomodados dentro de apretados estantes que están bajo la llave de uno, dos y hasta tres candados; mientras que los libros de las zonas accesibles se localizan en estantes abiertos y espaciosos para consultarlos rápidamente. En la cromatina, para silenciar la expresión de una o varias secuencias de adn, la célula agrega un grupo químico (o candado) llamado metilo a las histonas donde se encuentra empaquetado; las histonas pueden metilarse una, dos o hasta tres veces para silenciar. Cabe mencionar que no sólo la metilación regula la compactación del adn con las histonas, sino que hay otros grupos químicos que pueden actuar como llaves maestras para abrir o dar mayor accesibilidad al adn. Entre ellos, se encuentran principalmente la acetilación y la fosforilación, las cuales relajan la cromatina y facilitan el acceso de la maquinaria de transcripción al adn, activando de esta manera la expresión de los genes. A todas estas modificaciones se les conoce como marcas o modificaciones epigenéticas (Allis et al., 2015; Zhang et al., 2015; Henikoff y Greally 2016; ver figura 3).
Siguiendo con el ejemplo de la biblioteca, hay libros específicos cuya información requiere ser leída únicamente en un momento determinado del funcionamiento de la fábrica, por lo que se le pone un candado adicional para que sólo el personal autorizado lo abra cuando es necesario. En la célula, a medida que avanza la diferenciación celular, existen genes que necesitan silenciarse permanentemente por lo que, además de la metilación en las histonas, presentan metilación en su secuencia de nucleótidos en el adn, lo cual impide que las moléculas operadoras puedan leerlo (Allis et al., 2015; Bogdanović, y Lister, 2017; ver figura 3). La metilación del adn puede considerarse también como un candado epigenético. Otro candado se da por la relocalización de estos genes a distintas regiones en el núcleo, generalmente a la compacta periferia nuclear, lo que favorece que se mantengan silenciados. Ahora bien, no todos los genes que se metilan se silencian permanentemente, existen genes que únicamente tendrán algunos candados, por lo que podrían expresarse en respuesta a factores externos como por ejemplo la dieta o el ejercicio. Finalmente, otro mecanismo epigenético, generalmente implicado en silenciamiento, es el usado por pequeñas moléculas de nucleótidos de arn (ácido ribonucleico), que no codifican proteínas, por ejemplo, micrornas, sirnas, lncrnas y que difieren en su tamaño y mecanismo de acción (Morris y Mattick, 2014).
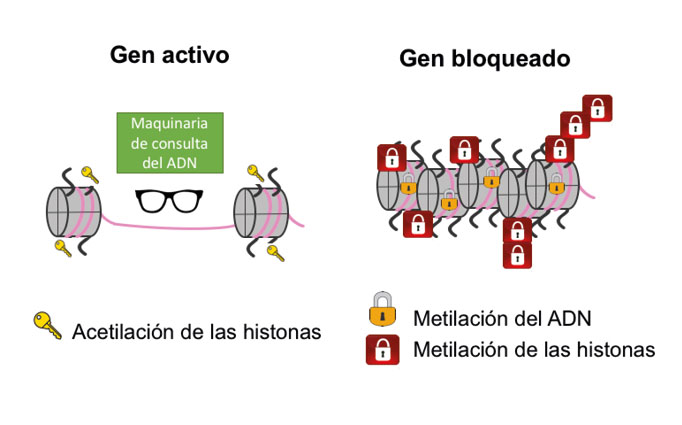
Figura 3. Las marcas epigenéticas pueden activar o bloquear la función de un gen. Un gen se activa cuando tiene grupos acetil en las histonas o se bloquea si tiene grupos metil en el adn y en las histonas. La maquinaria de consulta del adn representa el proceso de transcripción en la cromatina para que un gen pueda formar arn mensajero que dará lugar a una proteína con una función específica (Li et al., 2007; Skalska et al., 2017; Gottesfeld, 2019).
El ambiente modula el encendido y apagado de los genes
Existen diferentes moduladores que pueden provocar cambios epigenéticos en los organismos, entre ellos, el uso de fármacos, la interacción con otros organismos (por ejemplo, la simbiosis dada entre los humanos con su microbiota intestinal, o entre plantas y las rhizobacterias que fijan el nitrógeno del suelo), la exposición crónica a contaminación ambiental, cigarro y drogas; y el estilo de vida como la alimentación y el ejercicio (Dawson y Kouzarides, 2012; Miro-Blanch y Yanes, 2019; Asgari, 2014; Rider y Carlsten, 2019; Lötsch et al., 2013; Gao et al., 2016; Reid et al., 2017; Fernandes et al., 2017; McGee, 2019; ver figura 4).
En la naturaleza se presentan numerosos ejemplos de regulación epigenética. En las abejas, la diferencia entre la abeja reina y las obreras radica en la fertilidad. A pesar de que ambas provienen de larvas genéticamente iguales, la larva que será la reina recibe otro tipo de alimentación, que se basa en jalea real. La jalea real proporciona agentes que bloquean a la proteína dnmt3 (dna metil-transferasa). Esta proteína se encarga de metilar (agregar grupos metilo) al adn y, por lo tanto, de silenciar los genes relacionados con la morfología reproductiva en las abejas obreras, lo que impide que se reproduzcan (Kucharski, Maleszka, Foret y Maleszka, 2008).
La vernalización es un proceso epigenético, en el cual las plantas presentan una mayor floración en primavera si se exponen previamente a un invierno muy frío o prolongado. En la planta modelo Arabidopsis thaliana, y en otras como la uva (Vitis vinífera), existe un gen llamado flc (Flowering Locus C) que codifica para una proteína represora de la floración. En inviernos prolongados, este gen se encuentra con las histonas acetiladas que le permiten expresarse y cumplir la función de reprimir la floración; a la par también se expresan arns no codificantes específicos para el gen flc. Cuando la temperatura asciende, estas secuencias no codificantes del gen activan un mecanismo que trimetila las histonas de flc, lo cual pone candados a su expresión y permite, de esta manera, una buena floración en primavera. Es decir, este gen se autorregula mediante mecanismos epigenéticos en respuesta a la temperatura (Swiezewski et al., 2009; Feil y Fraga 2012).
Las llaves y candados en el desarrollo de los organismos y en la aparición de enfermedades
La regulación epigenética es crucial en todos los procesos del desarrollo de los seres vivos, ya que modula la expresión de genes que participan en la diferenciación, migración y proliferación celular, el desarrollo embrionario, el crecimiento y la reproducción (Atlasi y Stunnenberg, 2017). Cuando esta regulación es errónea en genes esenciales, se puede dar lugar a una mayor susceptibilidad al desarrollo de enfermedades (ver figura 4).
Durante el desarrollo embrionario se ha visto la participación de las marcas epigenéticas, las cuales pueden afectar la expresión de genes durante toda la vida. En los humanos es reconocida la influencia del estilo de vida y de la salud materna sobre el feto. Por ejemplo, los hijos de las mujeres que sufrieron violencia doméstica durante el embarazo tienen alterado el perfil de metilación del gen que codifica al receptor de cortisol, la hormona del estrés, lo cual se relaciona con problemas sociales de estos niños, como ansiedad, depresión, insomnio y ataques de pánico, principalmente durante la adolescencia (Radtke, et al., 2011).
Los cambios epigenéticos también influyen en el riesgo del desarrollo de enfermedades como artritis, Alzheimer, diabetes y cáncer (Hammaker y Firestein 2018; Wood 2018; Sommese, et al., 2018; Baylin y Jones 2016; Vicente-Dueñas, et al., 2018). Por ejemplo, en el cáncer de mama se ha visto un incremento de marcas represivas de metilación en el adn del gen brca1 (Breast Cancer 1), el cual produce una proteína supresora de tumores (Paydar, et al., 2019). Por el contrario, en cáncer de próstata se presenta un incremento de marcas de apertura de fosforilación en las histonas de genes blanco del ar (receptor de andrógenos), implicados en la proliferación celular (Kim, et al., 2014).
Sin embargo, se ha observado que el estilo de vida como la dieta puede modificar el patrón de candados y llaves de genes que regulan la aparición o el desarrollo de estas enfermedades. Por ejemplo, en la aparición de enfermedades, estudios epidemiológicos han mostrado que la alta ingesta de vegetales verdes esta inversamente relacionado con la aparición de diferentes tipos de cáncer (Giovannucci, et al., 2003; Egner 2014; Gerhauser 2013; Bishop y Ferguson, 2015). Aunado a lo anterior, se sabe que los productos químicos (metilos o acetilos) de la digestión de estos vegetales pueden funcionar como candados o llaves que cierran o abren genes importantes para el buen funcionamiento celular (Reid, et al., 2017; Li et al., 2018). Por otro lado, en lo que se refiere al desarrollo o progresión de enfermedades, se sabe que diversos fitoquímicos presentes en los vegetales y las frutas tienen efectos anticarcinogénicos dependientes del patrón de candados y llaves epigenéticas, en diferentes tipos de cáncer (Shankar, et al., 2016; Carlos-Reyes, et al., 2019). Por ejemplo, en cáncer de próstata, un compuesto químico contenido en el brócoli y la col de Bruselas incrementa las marcas de metilación o candados, en las histonas de genes implicados en la reparación del adn, bloqueando así́ su expresión lo que trae como consecuencia un aumento del daño al adn en la célula cancerosa. (Palomera-Sanchez, et al., 2017., ver figura 4).
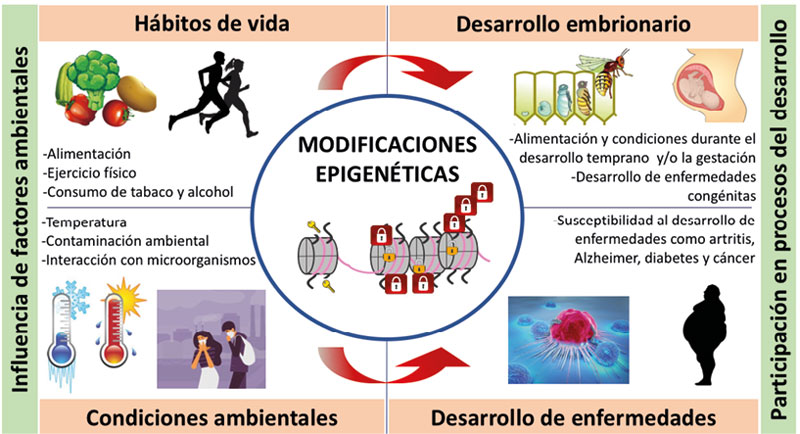
Figura 4. Los hábitos de vida y las condiciones ambientales modulan el desarrollo embrionario y la propensión a enfermedades, a través de las modificaciones epigenéticas. Por ejemplo, la alimentación durante la etapa larvaria de las abejas influye sobre su fertilidad; la temperatura ambiental influencia la floración de las plantas; el consumo de tabaco y drogas propicia el desarrollo de enfermedades; el ejercicio físico y la alimentación sana disminuyen la susceptibilidad a enfermedades como diabetes y cáncer. Todos estos procesos son regulados por las llaves (acetilaciones) y candados (metilaciones) que ponemos sobre nuestros genes.
Conclusiones
El estudio de los mecanismos epigenéticos nos ha permitido entender cómo el ambiente modula muchos fenómenos biológicos, por ejemplo, el fenotipo corporal y reproductivo, la floración y fruto de las plantas, el desarrollo embrionario, la aparición, desarrollo y prevención de enfermedades. De esta forma, a pesar de que no podemos cambiar nuestra secuencia de adn, sí podemos modificar la expresión génica mediante la dieta y el estilo de vida, gracias a que ahora contamos con una gran información, la de la epigenética, la cual puede influenciar positivamente la forma de regularla.
Referencias
- Allis, D., Caparros, ML., Jenuwein, T. y Reinberg, D. (2015). Epigenetics (2a ed.). Cold Spring Harbor Laboratory Press.
- Allis, D. y Jenuwein, T. (2016). The Molecular Hallmarks of Epigenetic Control. Nature Reviews Genetics, 17(8), 487-500. doi: https://doi.org/10.1038/nrg.2016.59.
- Asgari S. (2014). Epigenetic modifications underlying symbiont-host interactions. Advances in genetics, 86, 253–276. doi: https://doi.org/10.1016/b978-0-12-800222-3.00010-3.
- Atlasi, Y. y Stunnenberg, H. G. (2017). The interplay of epigenetic marks during stem cell differentiation and development. Nature Reviews Genetics, 18(11), 643-658. doi: https://doi.org/10.1038/nrg.2017.57.
- Baylin, S.B. y Jones, P. A. (2016). Epigenetic Determinants of Cancer [review]. Cold Spring Harbor Perspectives in Biolology, 8, a019505. doi: https://doi.org/10.1101/cshperspect.a019505.
- Bishop, K.S. y Ferguson, L.R. (2015). The Interaction between Epigenetics, Nutrition and the Development of Cancer. Nutrients, 7(2), 922-947. doi: https://doi.org/10.3390/nu7020922.
- Bogdanović, O. y Lister, R. (2017). dna Methylation and the Preservation of Cell Identity. Curr Opin Genet Dev., 46, 9-14. doi: https://doi.org/10.1016/j.gde.2017.06.007.
- Carlos-Reyes, A., López-González, J. S., Meneses-Flores, M., Gallardo-Rincón, D., Ruíz-García, E., Marchat, L. A. Astudillo-de la Vega, H., Hernández de la Cruz, O. N. y López-Camarillo, C. (2019). Dietary Compounds as Epigenetic Modulating Agents in Cancer. Front Genet, 10(79). doi: https://doi.org/10.3389/fgene.2019.00079.
- Dawson, M. A. y Kouzarides T. (2012). Cancer epigenetics: from mechanism to therapy. Cell, 150(1), 12-27. doi: https://doi.org/10.1016/j.cell.2012.06.013.
- Egner, P. E. (2014). Rapid and Sustainable Detoxication of Airborne Pollutants by Broccoli Sprout Beverage: Results of a Randomized Clinical Trial in China. Cancer Prevention Research, 7(8), 813-823. doi: https://doi.org/10.1158/1940-6207.CAPR-14-0103.
- Fraga, M. F., Ballestar, E., Paz, M. F., Ropero, S., Setien, F., Ballestar, M. L., Heine-Suñer, D., Cigudosa, J. C., Urioste, M., Benitez, J., Boix-Chornet, M., Sanchez-Aguilera, A., Ling, C., Carlsson, E., Poulsen, P., Vaag, A., Stephan, Z., Spector, T. D., Wu, Y., Plass, C. y Esteller, M. (2005). Epigenetic differences arise during the lifetime of monozygotic twins. pnas, 102(30), 10604-10609. doi: https://doi.org/10.1073/pnas.0500398102.
- Feil, R. y Fraga, M. F. (2012). Epigenetics and the environment: emerging patterns and implications. Nature Reviews Genetics, 13, 97–109. doi: https://doi.org/10.1038/nrg3142.
- Fernandes, J., Arida, R. M. y Gomez-Pinilla, F. (2017). Physical exercise as an epigenetic modulator of brain plasticity and cognition. Neuroscience and Biobehavioral Reviews, 80, 443-456. doi: https://doi.org/10.1016/j.neubiorev.2017.06.012.
- Gao, X., Zhang, Y., Breitling, L. P. y Brenner, H. (2016). Tobacco smoking and methylation of genes related to lung cancer development. Oncotarget, 7(37), 59017-59028. doi: https://doi.org/10.18632/oncotarget.10007.
- Garrett-Bakelman, F. E., Darshi, M., Green, S. J., Gur, R.C., Lin, L., Macias, B. R., et al. (2019). The nasa Twins Study: A Multidimensional Analysis of a Year-Long Human Spaceflight. Science, 364(6436). doi: https://doi.org/10.1126/science.aau8650.
- Gerhauser C. 2013. Epigenetic impact of dietary isothiocyanates in cancer chemoprevention. Curr Opin Clin Nutr Metab Care, 16(4), 405-10. doi: https://doi.org/10.1097/MCO.0b013e328362014e.
- Giovannucci, E., Rimm, E. B., Liu, Y., Stampfer, M. J. y Willett, W. C. (2003). A prospective study of cruciferous vegetables and prostate cancer. Cancer Epidemiol Biomarkers Prev, 12, 1403-1409.
- Gottesfeld, J. M. (2019). Milestones in Transcription and Chromatin Published in the Journal of Biological Chemistry. J Biol Chem, 294(5), 1652-1660. doi: https://doi.org/10.1074/jbc.TM118.004162.
- Hammaker, D. y Firestein, G. S. (2018). Epigenetics of Inflammatory Arthritis. Current Opinion Rheumatology, 30(2), 188-196. doi: https://doi.org/10.1097/BOR.0000000000000471.
- Henikoff, S. y Greally, J. M. (2016). Epigenetics, Cellular Memory and Gene Regulation. Current Biology, 26(14), R644-8. doi: https://doi.org/10.1016/j.cub.2016.06.011.
- Kim, J-Y., Banerjee, T., Vinckevicius, A., Luo, Q., Parkerm, J. B., Baker, M. R., Radhakrishnan, I., Wei, J. J., Barish, G. D. y Chakravarti, D. (2014). A Role for WDR5 in Integrating Threonine 11 Phosphorylation to Lysine 4 Methylation on Histone H3 During Androgen Signaling and in Prostate Cancer. Molecular Cell, 54(4), 613-25. doi: https://doi.org/10.1016/j.molcel.2014.03.043.
- Kucharski, R., Maleszka, J., Foret, S. y Maleszka, R. (2008). Nutritional Control of Reproductive Status in Honeybees via dna Methylation. Science, 319(5871), 1827-1830. doi: https://doi.org/10.1126/science.1153069.
- Li, B., Carey, M. y Workman, J. L. (2007). The Role of Chromatin During Transcription. Cell, 128(4), 707-19.
- Li, X., Egervari G., Wang Y., Berger, S. L. y Lu, Z. (2018). Regulation of Chromatin and Gene Expression by Metabolic Enzymes and Metabolites. Nature Reviews Molecular Cell Biology, 19(9), 563-578. doi: https://doi.org/10.1038/s41580-018-0029-7.
- Lötsch, J., Schneider, G., Reker, D., Parnham, M. J., Schneider, P., Geisslinger, G. y Doehring, A. (2013). Common non-epigenetic drugs as epigenetic modulators. Trends in Molecular Medicine, 19(12), 742-53 doi: https://doi.org/10.1016/j.molmed.2013.08.006.
- McGee, S. L. y Hargreaves, M. (2019). Epigenetics and Exercise. Endocrinol Metabolism, 30(9), 636-645. doi: https://doi.org/10.1016/j.tem.2019.06.002.
- Miro-Blanch, J. y Yanes, O. (2019). Epigenetic Regulation at the Interplay Between Gut Microbiota and Host Metabolism. Frontiers in genetics, 10, 638. doi: https://doi.org/10.3389/fgene.2019.00638.
- Morris, K. V. y Mattick, J. S. (2014). The rise of regulatory rna. Nature Reviews Genetics, 15(6), 423–437. doi: https://doi.org/10.1038/nrg3722.
- Palomera-Sanchez, Z., Watson, G. W., Wong, C. P., Beaver, L. M., Williams, D. E., Dashwood, R. H. y Ho, E. (2017). The Phytochemical 3,3’-diindolylmethane decreases expression of AR-controlled dna Damage Repair genes through repressive chromatin modifications and is associated with dna Damage in Prostate cancer cells. The Journal of Nutritional Biochemistry, 47, 113-119. doi: https://doi.org/10.1016/j.jnutbio.2017.05.005.
- Paydar, P., Asadikaram, G., Nejad, H. Z., Akbari, H., Abolhassani, M., Moazed, V., Nematollahi, M. H, Ebrahimi, G. y Fallah, H. (2019). Epigenetic modulation of brca1 and mgmt genes, and histones H4 and H3 are associated with breast tumors. Journal of Cellular Biochemistry, 120(8), 13726-13736. doi: https://doi.org/10.1002/jcb.28645.
- Radtke, K. M., Ruf, Gunter, H. M., Dohrmann, K., Schauer, M., Meyer, A. y Elbert, T. (2011). Transgenerational Impact of Intimate Partner Violence on Methylation in the Promoter of the Glucocorticoid Receptor. Translational Psychiatry, 1(7), e21. doi: https://doi.org/10.1038/tp.2011.21.
- Reid, M. A., Dai, Z. y Locasale, J. W. (2017). The Impact of Cellular Metabolism on Chromatin Dynamics and Epigenetics. Nature Cell Biology, 19(11), 1298-1306. doi: https://doi.org/10.1038/ncb3629.
- Rider, C. F. y Carlsten, C. (2019). Air pollution and dna methylation: effects of exposure in humans. Clinical Epigenetics, 11(1), 131. doi: https://doi.org/10.1186/s13148-019-0713-2.
- Shankar, E., Kanwal, R., Candamo, M. y Gupta, S. (2016). Dietary phytochemicals as epigenetic modifiers in cancer: Promise and challenges. Seminars in Cancer Biology, 40-41, 82-99. doi: https://doi.org/10.1016/j.semcancer.2016.04.002.
- Skalska, L., Beltran-Nebot, M., Ule, J. y Jenner, R. G. (2017). Regulatory Feedback From Nascent rna to Chromatin and Transcription. Nature Reviews Molecular Cell Bioly, 18(5), 331-337. doi: https://doi.org/10.1038/nrm.2017.12.
- Sommese, L., Benincasa, Lanza, M., Sorriento, A., Schiano, C., Lucchese, R., Alfano, R., Nicoletti, G. F. y Napolide, C. (2018). Novel Epigenetic-Sensitive Clinical Challenges Both in Type 1 and Type 2 Diabetes. Journal of Diabetes and its Complications, 32(11), 1076-1084. doi: https://doi.org/10.1016/j.jdiacomp.2018.08.012.
- Swiezewski, S., Liu, F., Magusin, A. y Dean, C. (2009). Cold-induced silencing by long antisense transcripts of an Arabidopsis Polycomb target. Nature, 462, 799–802. doi: https://doi.org/10.1038/nature08618
- Vicente-Dueñas, C., Hauer, J., Cobaleda, C., Borkhardt, A. y Sánchez-García, I. (2018). Epigenetic Priming in Cancer Initiation. Trends in Cancer, 4(6), 408-417. doi: https://doi.org/10.1016/j.trecan.2018.04.007.
- Waddington, C. H. (1942). The epigenotype. Endeavour, 1, 18–20.
- Waddington, C. H. (1942). Canalization of development and the inheritance of acquired characters. Nature, 150, 563–565.
- Wood, I. C. (2018). The Contribution and Therapeutic Potential of Epigenetic Modifications in Alzheimer’s Disease. Frontiers in Neurosciences, 12, 649. doi: https://doi.org/10.3389/fnins.2018.00649.
- Zhang, T., Cooper S. y Brockdorff, N. (2015). The Interplay of Histone Modifications – Writers That Read. embo Reports, 16(11), 1467-81. doi: https://doi.org/10.15252/embr.201540945.
Recepción: 01/02/2020. Aprobación: 31/08/2020.


